[All in One(2조)] 프로젝트로 배우는 데이터사이언스_3주차
탐색한 데이터로 모델성능 개선
[3주차]
작성일자: 2024-03-25
팀 구성원: 도우진, 오소민,오현정,정원준,최준헌
3. 1. 1 연속 수치 데이터를 범주형 변수로 변경하기
오버피팅(Overfitting)
: 머신러닝 모델이 학습 데이터에 과도하게 적합되어 새로운 데이터에 대한 예측 성능이 저하되는 현상
-> 수치의 범위가 넓으면 수치형 변수의 조건이 너무 세분화되어 tree가 깊어지므로 수치형 데이터를 범주화하여 성능을 높일 필요가 있음.
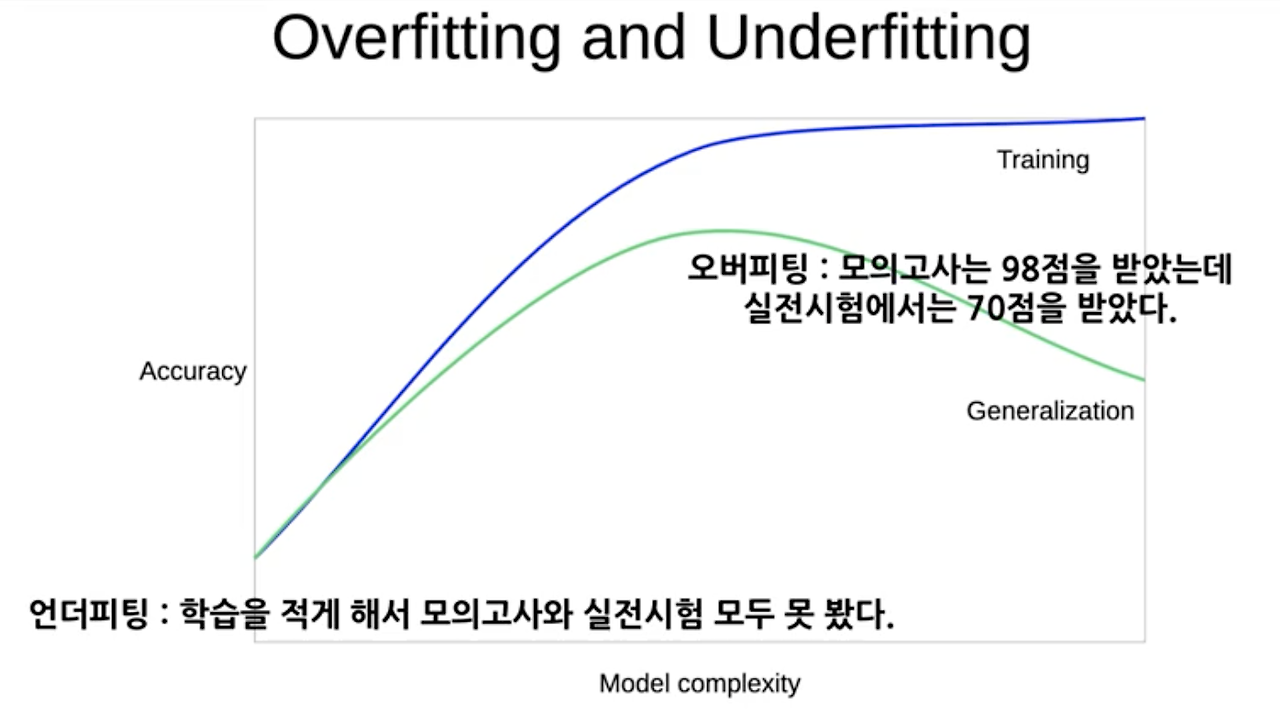
Feature engineering
: 머신러닝 모델의 성능을 향상시키기 위해 사용되는 데이터 전처리 과정
-> 오버피팅을 방지하고 모델의 성능을 향상시킴.
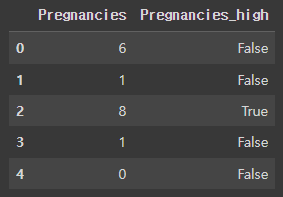
1. 수치형 변수를 범주형 변수로 만들기
df["Pregnancies_high"] = df["Pregnancies"] > 6
df[["Pregnancies", "Pregnancies_high"]].head()임신 횟수가 7회가 넘어가면 대체적으로 발병수가 높다는 사실을 저번에 알아보았기 때문에 임신 횟수가 7회 이상인 것을 Pregnancies_high로 지정.
2. 학습, 예측에 사용할 컬럼
feature_names = train.columns.tolist()
feature_names.remove("Pregnancies")
feature_names.remove("Outcome")
feature_namesPregnancies와 Outcome을 제외한 다른 feature들을 리스트 형태로 만들어 준다.
3. 정답값이자 예측해야 할 값
label_name = 'Outcome'
label_name'Outcome'Outcome을 분류하고 싶은 답안으로 지정한다.
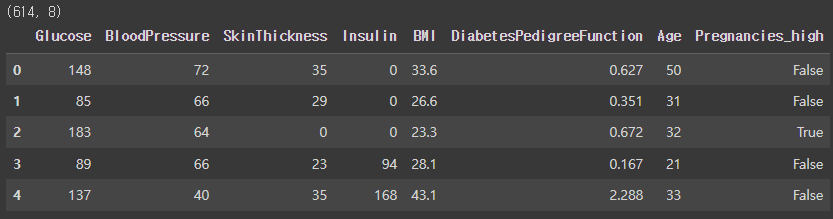
4. 학습, 예측 데이터셋 만들기
X_train = train[feature_names]
print(X_train.shape)
X_train.head()학습에 사용할 데이터셋을 만들어준다.
y_train = train[label_name]
print(y_train.shape)
y_train.head()학습 데이터셋의 정답 값을 만들어준다.
X_test = test[feature_names]
print(X_test.shape)
X_test.head()예측에 사용할 데이터셋을 만들어준다.
y_test = test[label_name]
print(y_test.shape)
y_test.head()예측 데이터셋의 정답 값을 만들어준다.
5. 머신러닝 알고리즘 가져오기
from sklearn.tree import DecisionTreeClassifier
model = DecisionTreeClassifier(random_state=42)
model머신러닝 알고리즘을 가져온다.
6. 학습(훈련) 하기
model.fit(X_train, y_train)학습 데이터셋과 정답 값을 통해서 학습을 한다.
7. 예측하기
y_predict = model.predict(X_test)
y_predict[:5]예측 데이터셋을 토대로 정답을 예측한다.

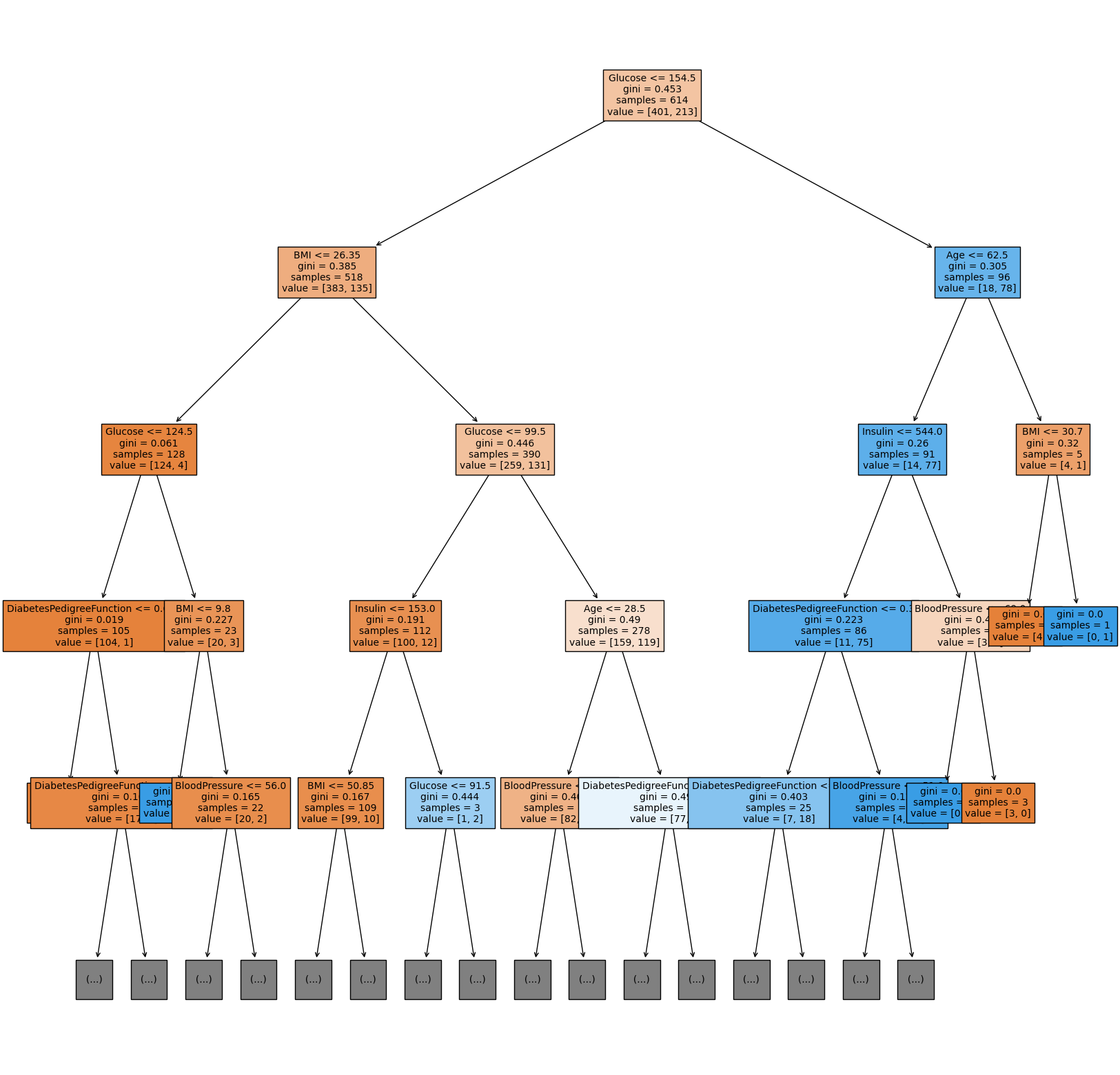
8. 트리 알고리즘 분석하기
from sklearn.tree import plot_tree
plt.figure(figsize=(20, 20))
tree = plot_tree(model,
feature_names=feature_names,
max_depth=4,
filled=True,
fontsize=10)의사결정나무를 시각화한다. 시각화된 의사결정나무를 살펴보면, Glucose가 가장 중요한 역할을 한다는 사실을 알 수 있다.
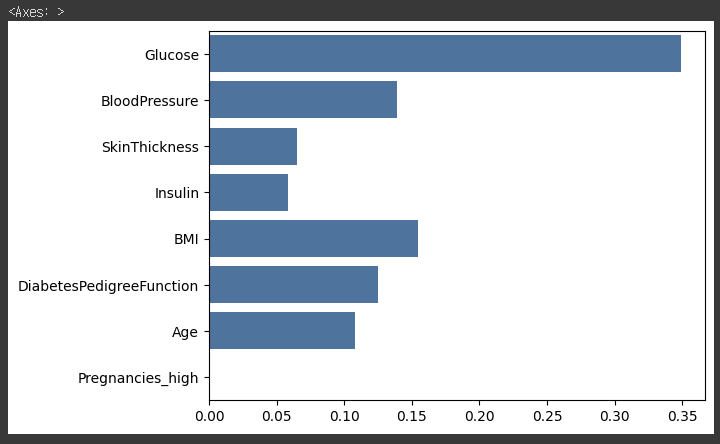
9. 피쳐의 중요도 시각화하기
sns.barplot(x=model.feature_importances_, y=feature_names)피쳐의 중요도를 시각화한 결과, 이전과는 다르게 Pregnancies가 중요한 역할을 하지 않고, Glucose가 가장 중요한 역할을 한다.
10. 정확도 측정하기
diff_count = abs(y_test - y_predict).sum()
diff_count39틀린 개수가 44개에서 39개로 줄어듦.
(len(y_test) - diff_count) / len(y_test) * 10074.67532467532467예측의 정확도도 기존의 71점에서 74점으로 좋아짐.
3.1.2 범주형 변수를 수치형 변수로 변환하기
지난 학습 Age를 Outcome에 따라 EDA 탐색을 했을때, distplot 분포도는 이렇게 나왔다.
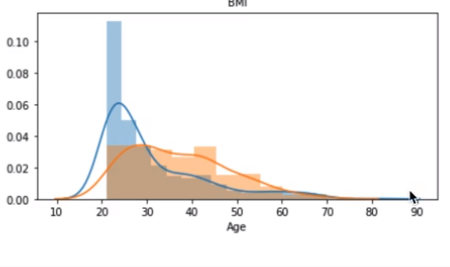
Age 구간에 따라 모델 성능도 달라질 수 있다. 따라서, 나이의 범위를 25세미만, 25세~60세미만, 60세 초과로 나눠보려고 한다.
#age 나눠보기
#나이가 25세미만, 25~60세, 60세초과으로 만들어보기
#3개의 카테고리로 나눠보기
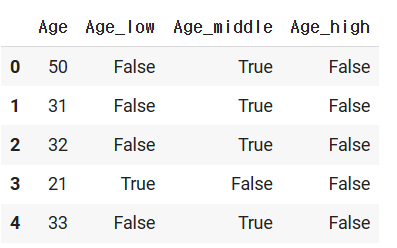
df['Age_low']=df["Age"]<25
df['Age_middle']=(df["Age"] >=25) & (df['Age']<=60)
df["Age_high"]=df['Age']>60
df[['Age','Age_low','Age_middle',"Age_high"]].head()25세미만은 Age_low열로, 25세~60세는 Age_middle로, 60세 초과는 Age_high로 만들어 주었다.
값들은 boolean 값들로 구성되었으며, 이것은 기계학습이 더 학습하기 쉬운 형태로 알려주는 것이다.
One-Hot-Encoding은 범주형 변수를 이진 벡터로 표현하는 것이고, 각 범주는 해당 범주에 해당하는 인덱스만 1이고 나머지는 0으로 표현된다. 강의에서는 One-Hot-Encoding에 대해 굉장히 모호하게 설명했는데, 내가 이해한 바에 따르면 저런 True,False 값들을 0,1 여부로 바꾸면,OneHotEncoding 되었다고 볼 수 있을 것 같다.
추가로, One-Hot-Encoding의 사용법을 찾아보았다. 직접 이렇게 하나하나씩 바꿔줘도 되지만, sklearn.processing에는 OneHotEncoder라는 알고리즘을 제공한다.
모델 객체를 정의하는 것과 같이 객체로 OneHotEncoder를 저장해주고, 데이터 프레임을 fit 시킨 후, transform 함수로 바꿔주면 된다. 그럼 아래와 같이 array 객체가 나올 것이다. 이렇게 Encoding 한 것을 df로 나타내기 위해선 각 열의 이름을 지정해줘야하는데 get_feature_names_out()이라는 함수를 써주면 특징 값들을 뽑아준다.
from sklearn.processing import OneHotEncoder
encoder=OneHotEncoder(sparse=False)
encoder.fit(df)
encod_array=encoder.transform(df)
#DataFrame으로 변환
pd.DataFrame(encod_array.astype(int),columns=encoder.get_feature_names_out())<예시>
위와 같이 프로세스 (학습 +예측 데이터셋 나누기 및 기계학습)을 해주면, Age_middle이 학습에 중요한 Feature로 작용했음을 알 수 있다.

다만, 결과를 해석해보면 diff_count 갯수가 오히려 늘어났다. 확연한 구분이 되지 않은 특징들이 많으면 오히려 정확도가 떨어질수 있다고 한다. Age 값을 나누면 성능이 더 안 좋게 나오므로, 이 피처엔지니어링 방법을 택하지 않는것이 좋겠다.
3.1.3 결측치 평균값으로 대체하기
결측치(NaN)가 들어있는 행을 drop 해주는 방법도 있지만, 이 방법이 항상 옳은 것은 아니다.
때론 결측치를 평균값 혹은 중앙값으로도 채워 줄 수 있다. 이번에는 결측치를 평균값으로 대체하는 방법을 학습해보려고 한다.
df.isnull().sum()을 하면 NaN은 보이지 않는다.
※ isnull(): 결측치가 있는지 물어보고 boolean값을 도출해준다.
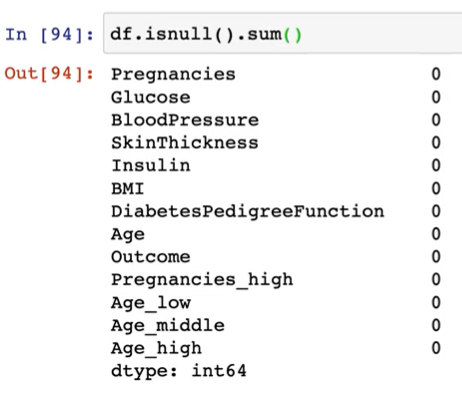
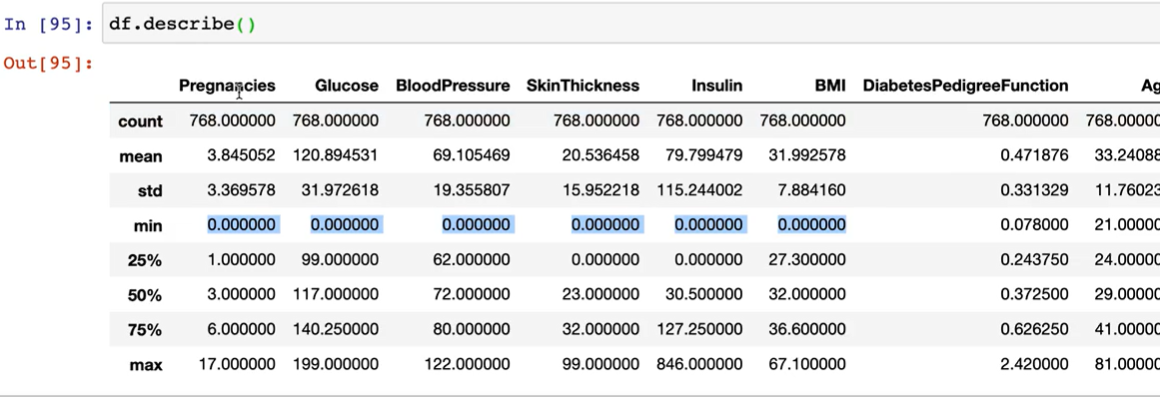
이럴때는 describe() 함수를 해서 각 열의 요약본을 봐보자. min 값들을 살펴보면, Pregnancies(임신횟수)는 0일수 있다고 쳐도, Glucose나 BloodPressure,SkinThickness,Insulin 등등은 0이 될 수 없다. 결측치는 없어도 때론, 0 값이 무의미한 값들이 될수 있다.
그 중 이번 학습에서는 Insulin 값들을 자세히 살펴보려고 한다. 2주차 학습 EDA 탐색하기 에서 boxplot으로 시각화해본 결과 인슐린 수치에는 결측치가 많음을 확인했다. 그래서 0보다 큰 값에 대해서만 그래프를 그렸던 것이었다.
sns.boxplot(data=df[df['Insulin']>0],x='Pregnancies,y='Insulin',hue='Outcome')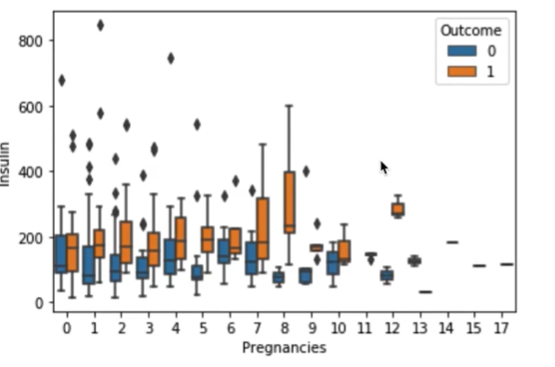
자 그럼 Insulin이 0인 값을 NaN으로 먼저 바꿔주고, 이 값에 평균값을 넣어주는 작업을 해보자.
1. Insulin 값이 0인 곳에 NaN 처리를 해주고, 이를 Insulin_nan이라는 새로운 열에 추가하기
# Insulin 열에서 0인 값들을 np.nan으로 처리해주자
df["Insulin_nan"]=df["Insulin"].replace(0,np.nan)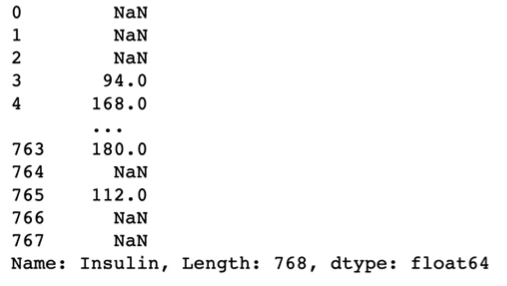
#결측치의 차지하는 비율
df['Insulin_nan'].isnull.mean()결측치가 차지하는 비율을 보면 약 48%이다. 우리는 이 결측치를 꼭 처리해줘야한다.
2. Outcome 여부에 따라 Insulin과 Insulin_nan의 차이를 살펴보자
#결과값에 따라 Insulin과 Insulin_nan의 평균값을 구해보자
df.groupby(['Outcome'])[["Insulin",'Insulin_nan']].mean()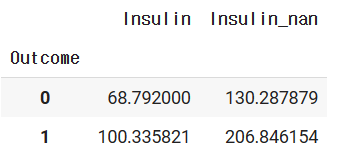
NaN 처리를 해줬을때랑 안 했을때 차이가 많이 나는 것을 알 수 있다. 우리의 최종 목표는 기계 학습을 시키는 것인데, NaN값이 존재하기 때문에 학습하려고 하면 Value Error가 발생한다. 따라서, 결측치를 채워 줘야한다.
3. Outcome 여부에 따라 Insulin과 Insulin_nan의 평균값과 중앙값을 구해보자
df.groupby(['Outcome'])[["Insulin",'Insulin_nan']].agg(['mean','median'])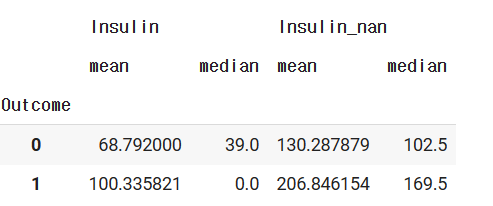
Outcome이 0 일때는 mean 값이 약 130, Outcome이 1 일때는 mean 값이 약 206임을 알 수 있다.
4. 결측치를 다음 평균값으로 이제 채워주자
#결측치 채우기
#Outcome에 때라 평균값이 다르니까
#Outcome이 1일때와 0일때 각각 채워주
df.loc[(df["Outcome"]==1) & (df["Insulin_nan"].isnull()),"Insulin_nan"]=206
df.loc[(df["Outcome"]==0) & (df["Insulin_nan"].isnull()),"Insulin_nan"]=130
5. 학습 모델 성능이 얼마나 높아졌는지 알아보자

1) 트리 시각화에서 Insulin_nan 열 값이 학습에 중요한 feature 역할을 했다는 걸 알 수 있다.
2) diff_count수가 줄어들었다.
3) 모델 성능이 85%를 달성 하였다.
따라서 결측치를 바꿔주는 작업은 꽤 모델의 성능을 높여줌을 이번 학습을 통해 배웠다.
3.1.4 결측치를 중앙값으로 대체하기
이번에는 Insulin의 결측치를 중앙값으로 대체해보자.
df.groupby(["Outcome"])[["Insulin", "Insulin_nan"]].agg(["mean", "median"])
우선 groupby() 함수를 이용하여 Outcome에 따른 결측치를 제거 / 제거하지 않은 Insulin의 평균과 중앙값을 구해보았다.
결과 :
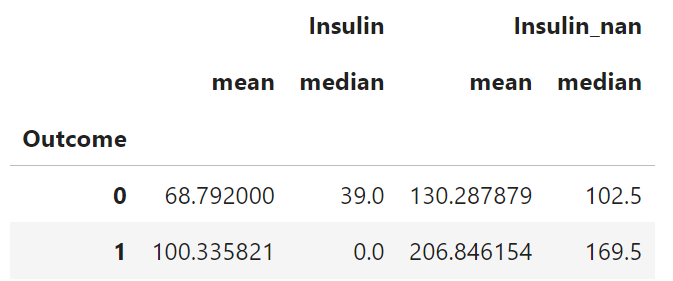
우리는 결측치가 포함된 Insulin_nan 데이터를 쓸 것이기에 그 부분에 주목해 보자. 당뇨병이 발병하지 않을 때(Outcome=0) Insulin_nan의 중앙값은 102.5, 당뇨병이 발병할 때(Outcome=1) Insulin_nan의 중앙값은 169.5이다.
df.loc[(df["Outcome"] == 0) & (df["Insulin_nan"].isnull()), "Insulin_nan"] = 102.5
df.loc[(df["Outcome"] == 1) & (df["Insulin_nan"].isnull()), "Insulin_nan"] = 169.5
위의 코드는 Outcome = 0으로 도출되는 Insulin_nan의 결측치를 102.5로, Outcome = 1로 도출되는 Insulin_nan의 결측치를 169.5로 채운 것이다. 그 후 학습과 분류를 똑같이 해준 후, plot tree를 그려보았다.
plt.figure(figsize=(20,20))
tree = plot_tree(model, feature_names=feature_names,
filled=True, fontsize=10)
결과 :
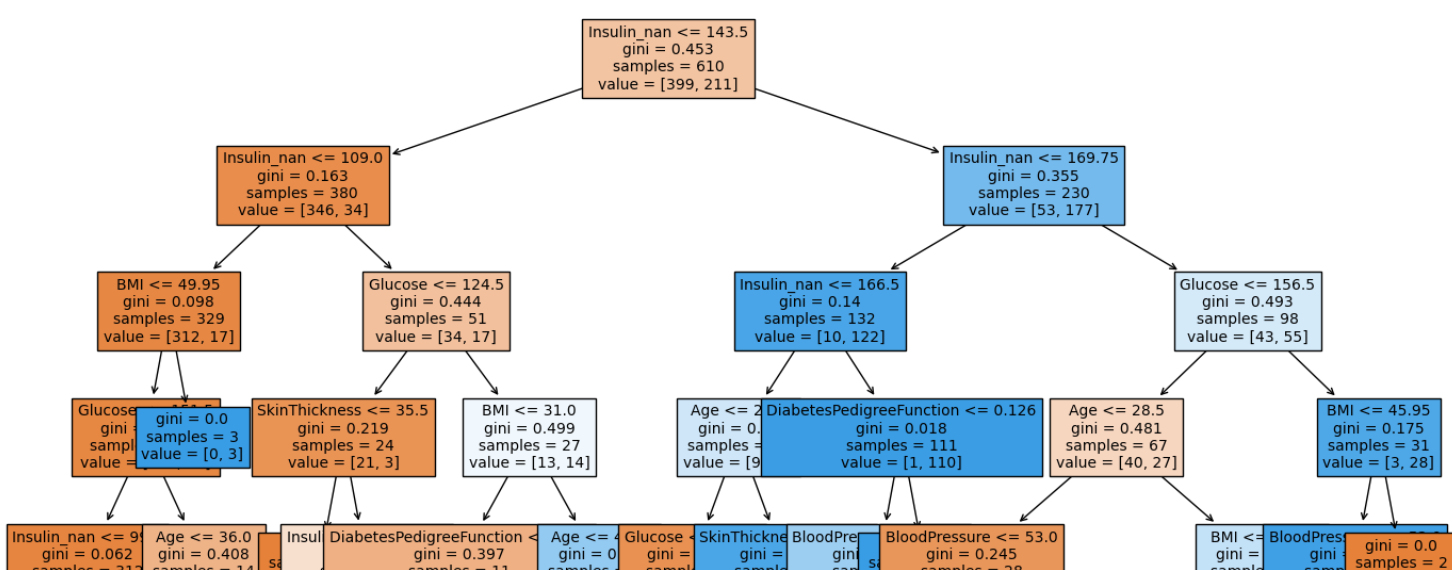
Insulin_nan 데이터의 중요도가 올라간 것으로 보인다. 정확하게 판단하기 위해 feature importance를 구해서 시각화해보자.
sns.barplot(x=model.feature_importances_, y=feature_names)
결과:
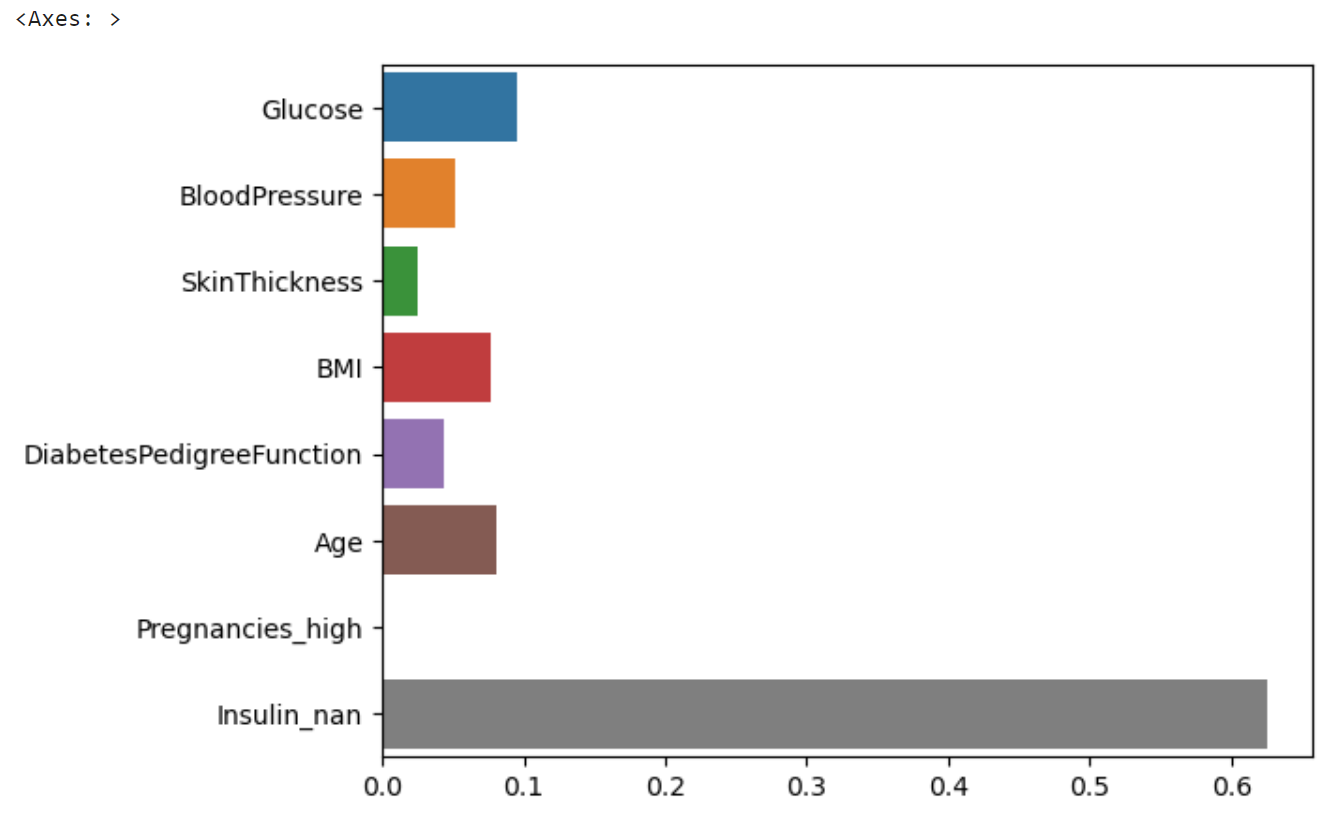
Insulin_nan의 중요도가 가장 높은 것을 확인할 수 있다. 모델의 정확도도 확인해보자.
diff_count = abs(y_test - y_predict).sum()
diff_count
17
가장 적은 오차가 나왔다. 결측치에 평균을 채워넣은 모델보다 성능이 좋은 것을 확인할 수 있다.
(len(y_test) - diff_count) / len(y_test) *100
88.96103896103897
대량의 결측치를 값으로 바꾸어주었더니 나타난 결과이다. 이처럼 결측치가 많을 경우 제거하기 보다는 적절한 수로 치환하는 것이 전처리의 한 방법이다.
3.1.5 수치형 변수를 정규분포 형태로 만들기
모델의 성능을 개선하는 다른 방법을 알아보자. 우선, 0보다 큰 Insulin의 값을 distplot을 통해 그려본다.
sns.distplot(df.loc[df["Insulin"] > 0,"Insulin"])
결과:
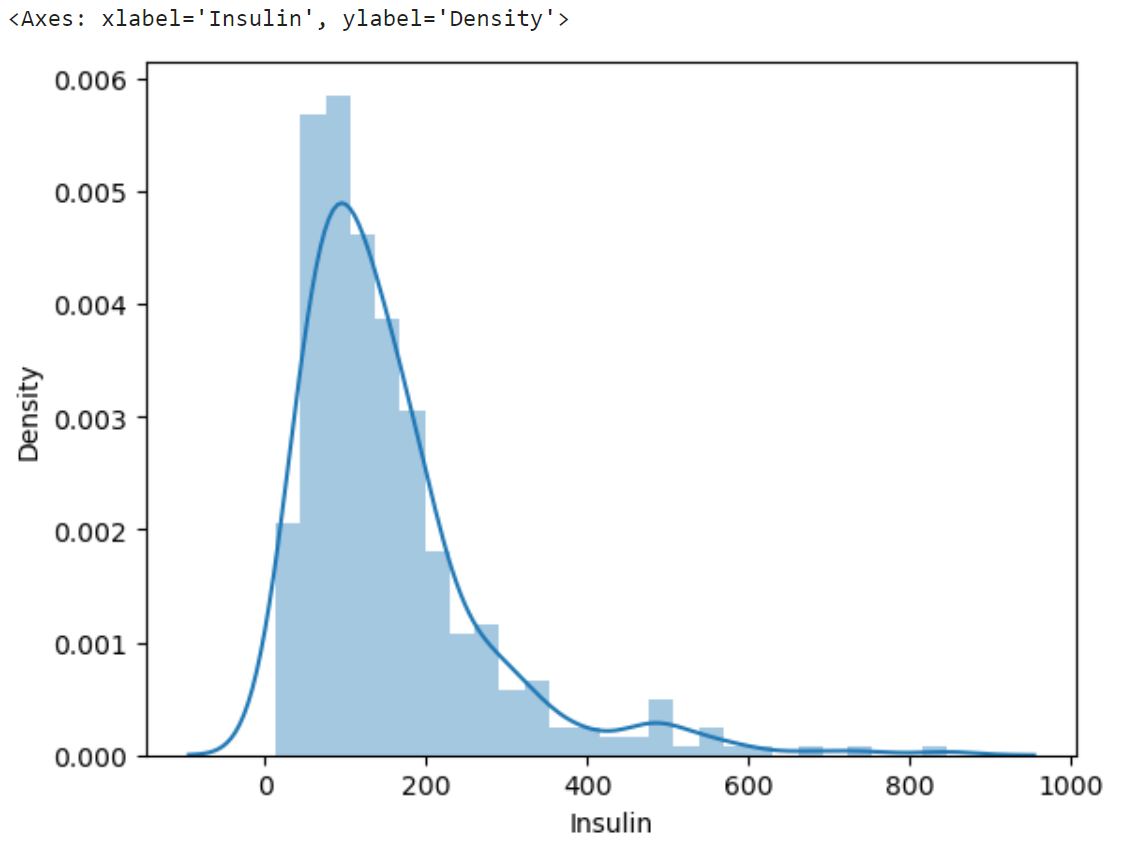
'왜도'란 한쪽으로 치우친 정도를 의미하고, '첨도'란 뾰족한 정도를 의미한다.
위에서 보다시피 현재 Insulin 데이터는 왜도와 첨도가 높은 차트이다. 이렇게 데이터가 몰려있으면 학습에 어려움이 생긴다. 따라서 정규분포 모양으로 만들어보려고 한다.
sns.distplot(np.log(df.loc[df["Insulin"] > 0,"Insulin"] + 1))
log 변환을 하면 정규분포의 형태를 띠게 만들 수 있다. 그러나 로그함수는 0이하에서는 음의 무한대 값으로 수렴하므로 +1을 해준다.
결과 :
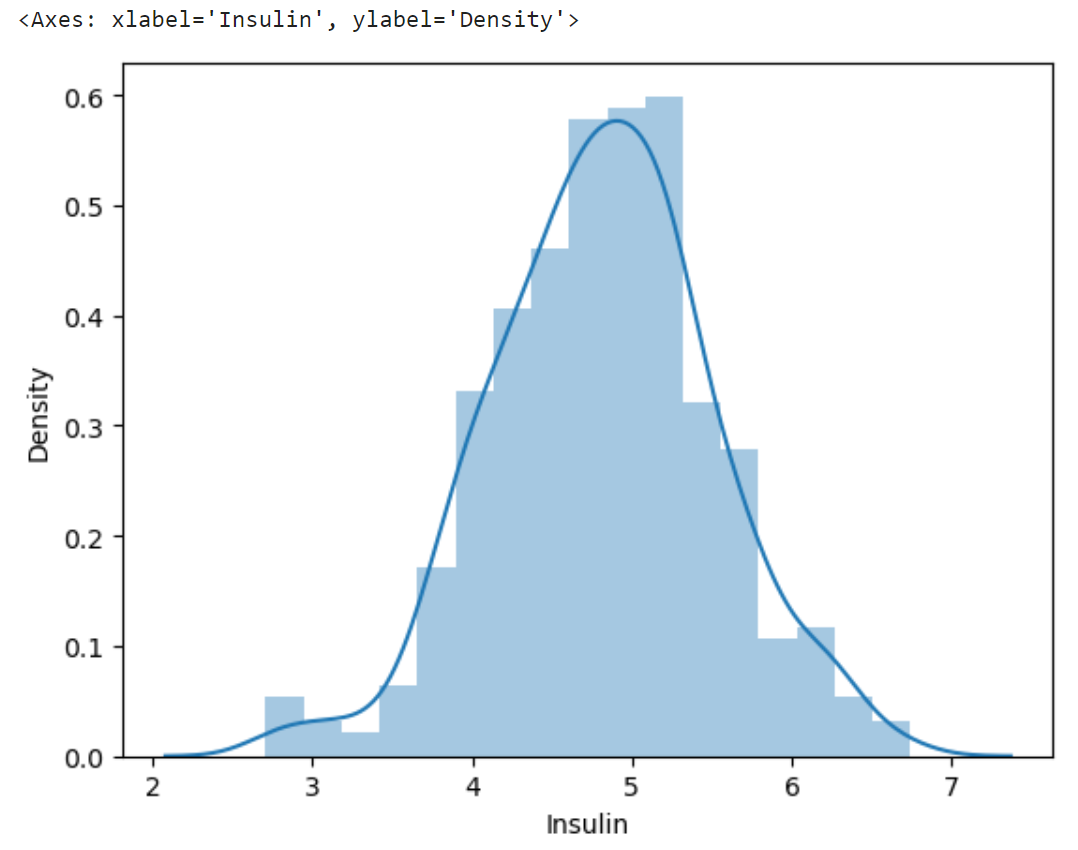
직접 모델 학습에 사용할 Insulin_nan 데이터에도 적용해보자.
sns.distplot(df["Insulin_nan"])
대량의 결측치를 중앙값으로 채워줬기 때문에 값이 뾰족하게 튀어나와있다.
결과 :
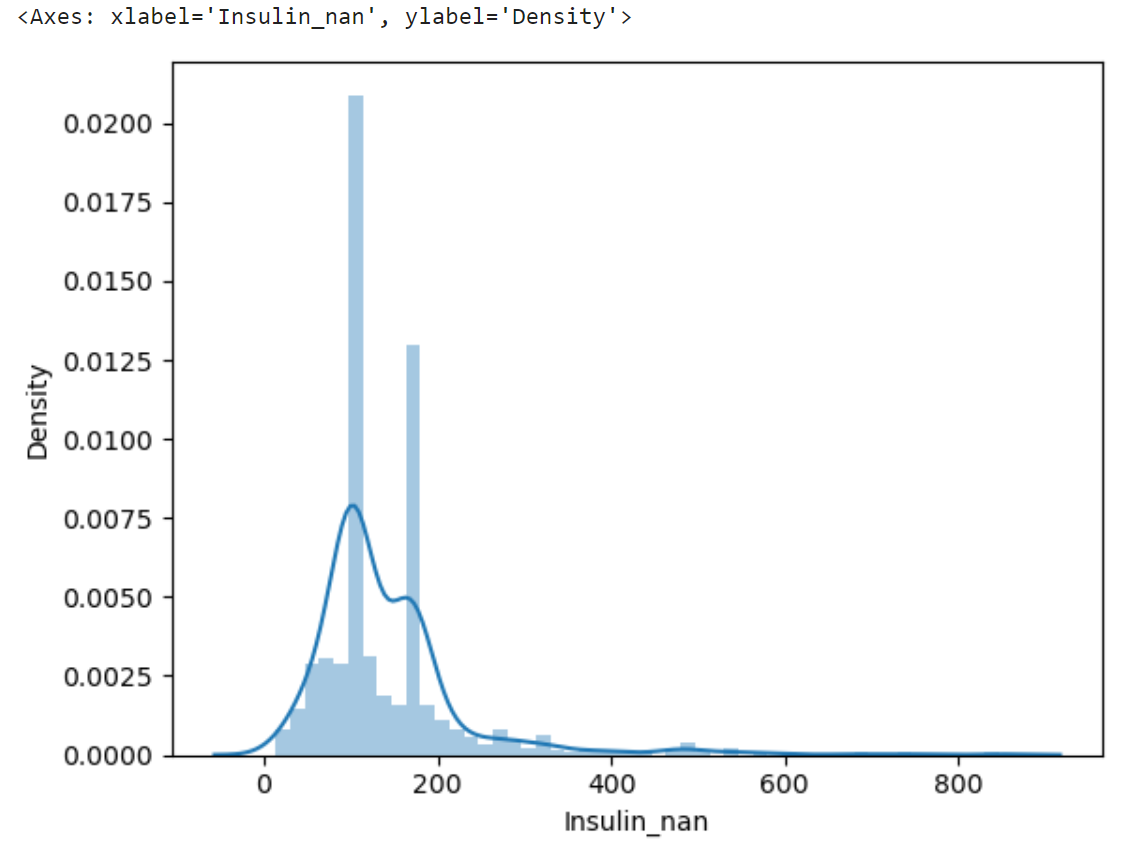
log 변환을 하여 그 값을 Insulin_log라는 변수에 저장한다. 똑같이 log 변환 후의 distplot으로 그려보자.
df["Insulin_log"] = np.log(df["Insulin_nan"] +1)
sns.distplot(df["Insulin_log"])
결과 :
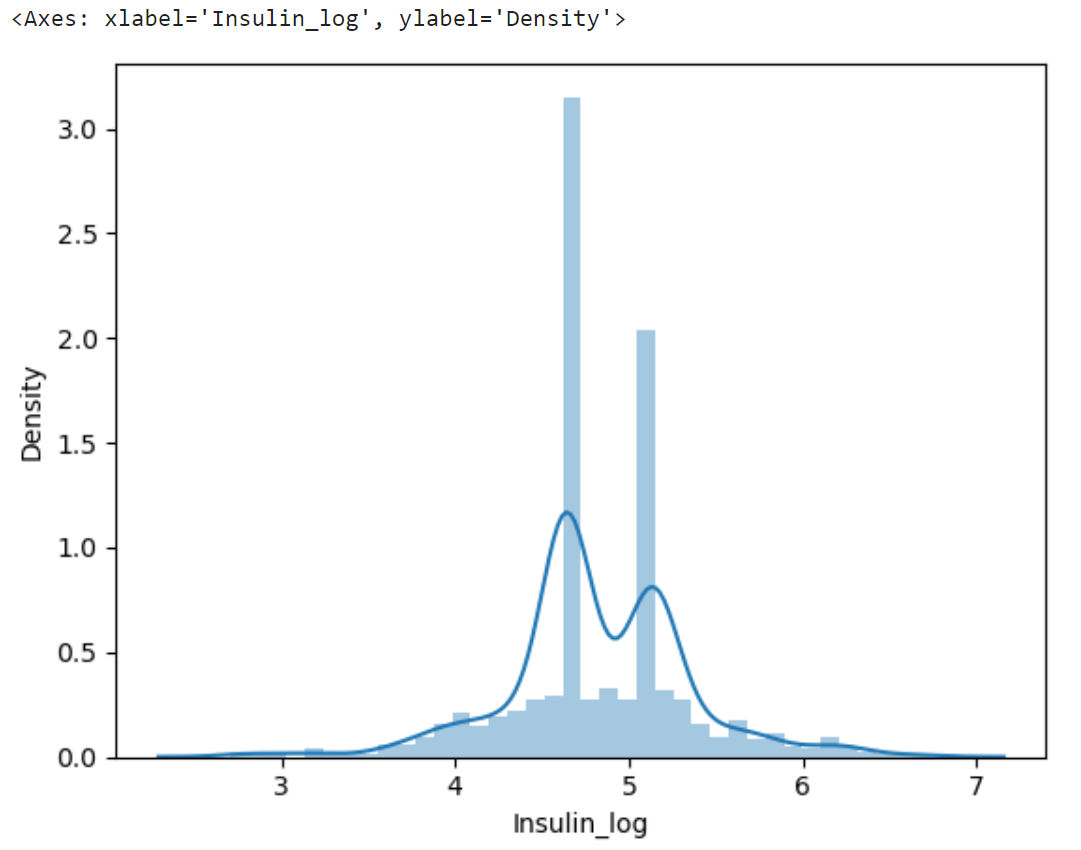
원래보다 왜도와 첨도가 낮은 차트가 그려진다. 이제 Insulin_nan 대신 Insulin_log를 넣어 모델 학습과 예측을 해본다.
결과 :
diff_count = abs(y_test - y_predict).sum()
diff_count
20(len(y_test) - diff_count) / len(y_test) *100
87.01298701298701
그러나 log 변환을 하기 전 데이터를 넣은 모델보다 오차가 크고, 정확도가 떨어진 결과를 보여준다. log 변환을 하면 보통 더 나은 성능을 보이지만, 이 데이터에서는 변화가 없는 모습이다. 따라서 이 데이터셋에 한해서는 log 변환을 하지 않은 데이터를 넣은 모델이 성능이 좋다는 사실을 파악할 수 있다.
3.1.6 상관 분석을 통해 파생변수 만들기
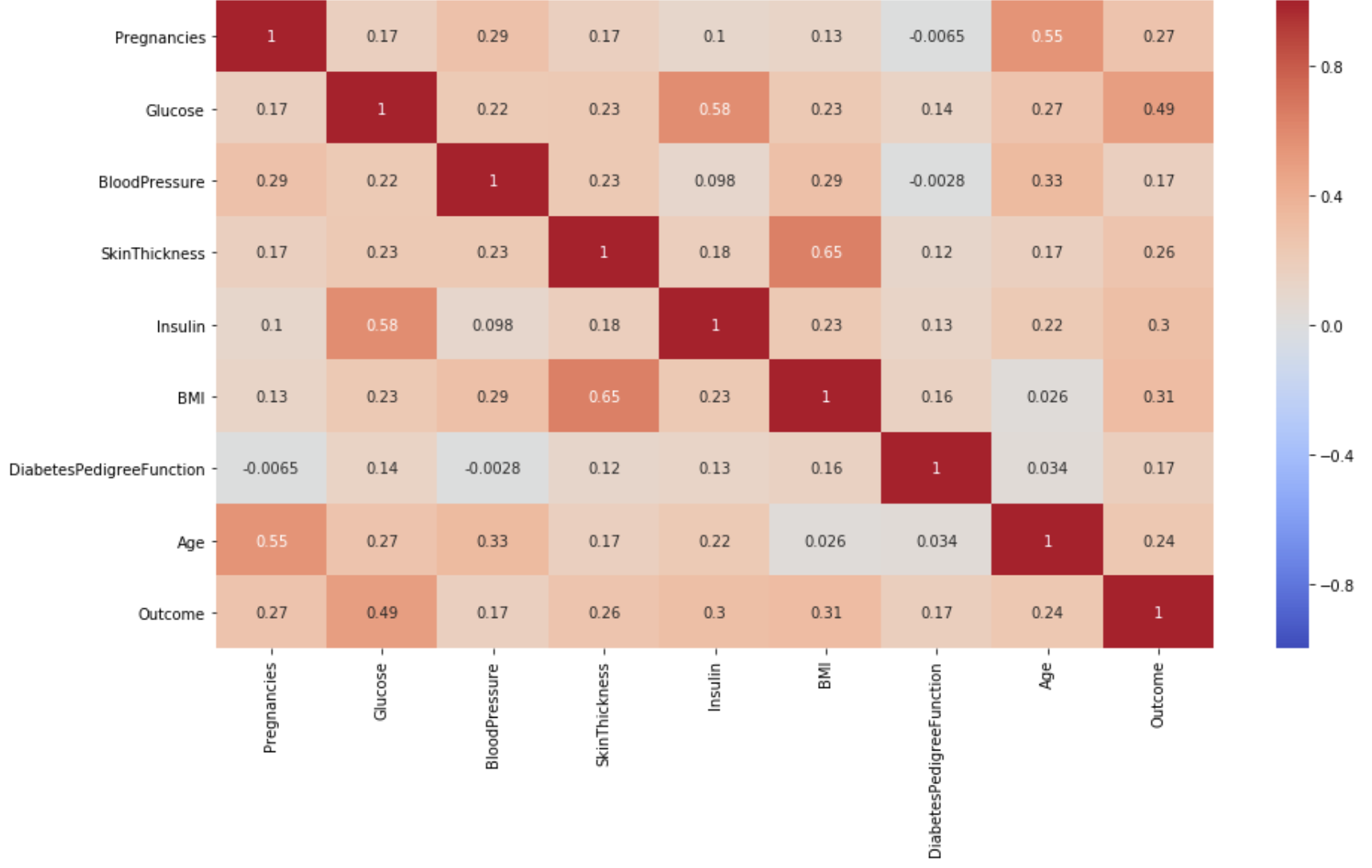
이것은 각 컬럼별 상관계수를 나타낸 히트맵이다. 이 중에서 인슐린과 글루코스의 상관계수, BMI와 피부의 두께 정도 간의 상관관계가 0.65로 다른 컬럼보다 더 높은 값을 가지고 있는 것을 알 수 있다.
여기서 regplot을 이용해 인슐린과 글루코스, BMI와 피부두께정도(Skinthickness) 의 그래프를 보도록 하자.
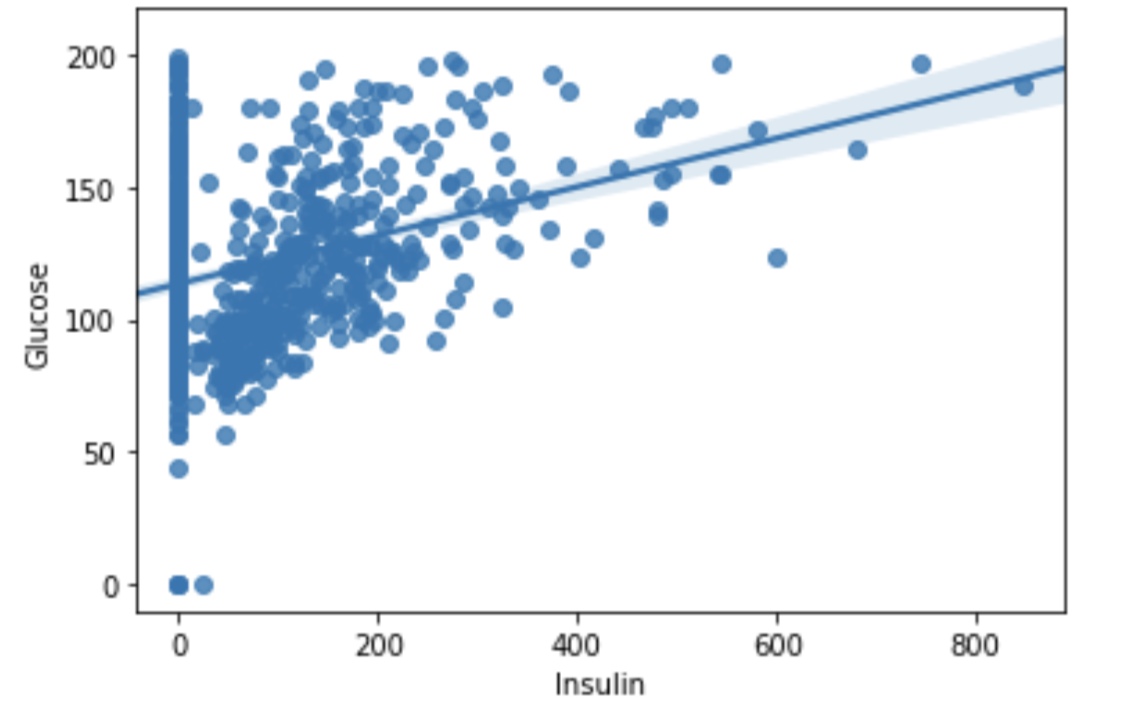
먼저 글루코스와 인슐린의 pairplot을 보면 인슐린이 0일때의 값이 몰려있는 것을 알 수 있는데, 이로 인해 추세선의 값에 영향을 준다. 그래서 인슐린 값이 0인 Num값을 제거해주고 pairplot을 다시 출력해보자.
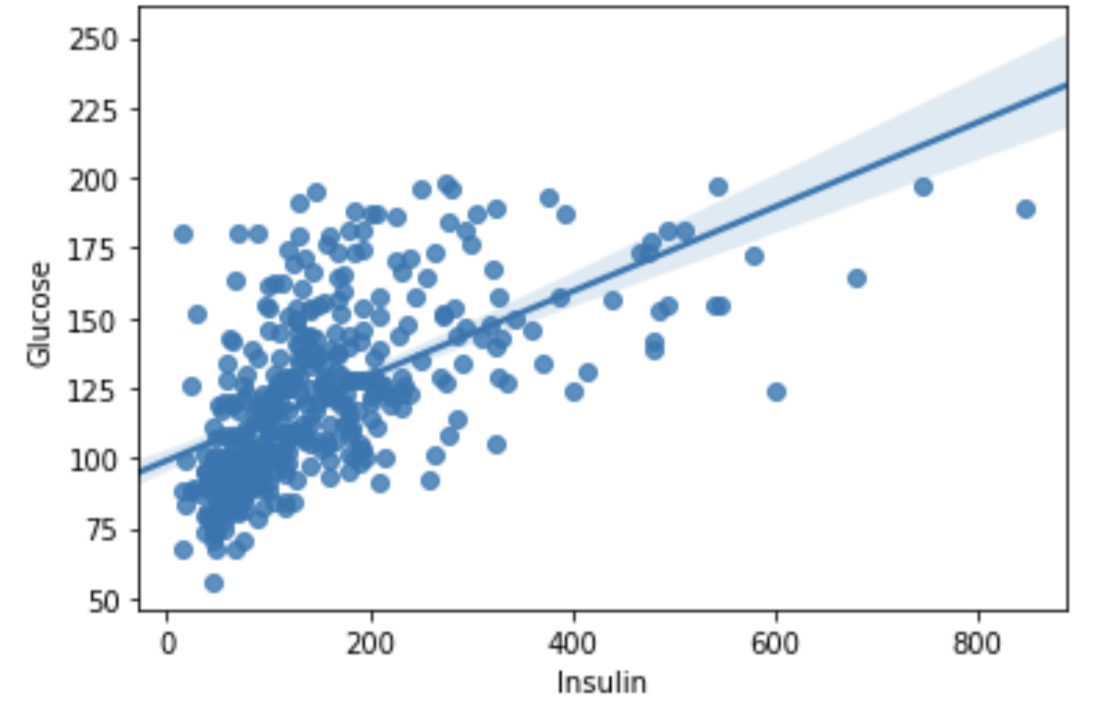
그 결과 인슐린과 글루코스 간의 산점도 그래프를 통해 양의 상관관계가 있음을 유추해낼 수 있다.
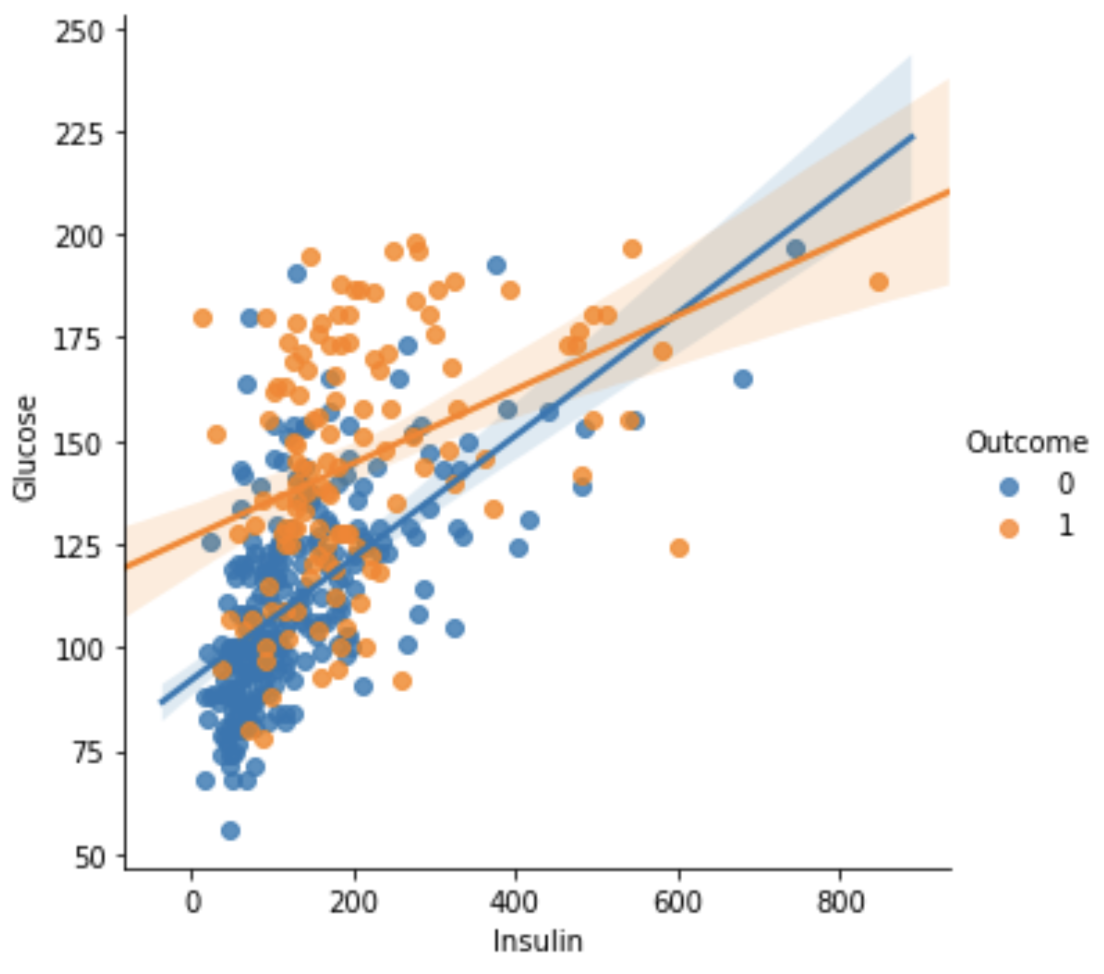
글루코스 값이 100 미만, 인슐린이 102.5 이하일 때 outcome이 0 인. 즉, 당뇨병이 발병하지 않는 케이스가 몰려있는 것을 확인할 수 있다.
그럼 이 상황을 좀 더 자세히 파헤쳐보기 위해서 글루코스 값이 100보다 작고, 인슐린의 값이 102.5 이하인 것을 다른 변수로 설정해보자.
df["low_glu_insulin"] = (df["Glucose"] < 100) & (df["Insulin_nan"] <= 102.5)
df["low_glu_insulin"].head()pd.crosstab(df["Outcome"], df["low_glu_insulin"])당뇨병 발병 여부와 낮은 글루코스, 인슐린의 관계를 알아보기 위해 데이터프레임을 출력해보자.

이런 데이터 프레임을 얻을 수 있는데, False는 low glucose, insulin이 아닌 경우, True인 경우는 low glucose, insulin 을 가질 경우에 대해 당뇨병 발병 여부를 나타낸 것이다.
그러니 이 데이터에서는 low glucose와 insulin의 상태일 때 당뇨병이 발병한 케이스는 전체 768건 중 5건으로, 발병확률이 매우 낮다고 할 수 있겠다.
이러한 파생변수를 만들고 난 뒤 다시 모델을 학습시켜보자.
모델을 학습 시키는 과정은 1장에서 배운 기존 방법과 같으므로 생략함.
인슐린 결측치를 중앙값으로, 낮은 인슐린 글루코스 파생변수를 학습에 추가했고, outlier 값도 제거한 결과가 나오게 된다.
diff_count = abs(y_test - y_predict).sum()
diff_countdiff_count에는 y_test와 y_predict 사이의 차이의 절대값의 총합이 저장된다. 예측 값과 실제 값 사이의 차이의 절대값이 총 15개의 데이터 포인트에 대해 계산한다. 이후 직접 정확도를 구하는 식에 diff_count를 넣어 정확도를 계산할 수도 있고, 아래 코드 처럼 정확도를 계산하는 알고리즘을 가져올 수도 있다.
from sklearn.metrics import accuracy_score
accuracy_score(y_test, y_predict) * 100model.score(X_test, y_test) * 100이 정확도를 계산한 결과 90.25974025974025의 값이 나왔으므로 모델의 학습 정도가 매우 뛰어난 것을 알 수 있다.
3.1.7 이상치 다루기
여기서 다루는 이상치란 boxplot을 통해 중앙값에서 멀리 떨어진 결측치를 제거하여 자료의 신뢰성과 정확도를 높이는 방법이다.
인슐린 값이 중앙값에 비해 너무 큰 값을 가지는 것을 찾아내기 위해 boxplot을 출력해보자.
plt.figure(figsize=(15, 2))
sns.boxplot(data=df, x="Insulin_nan")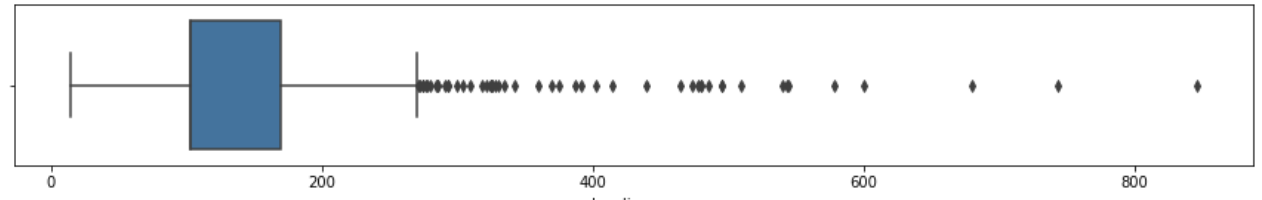
df["Insulin_nan"].describe()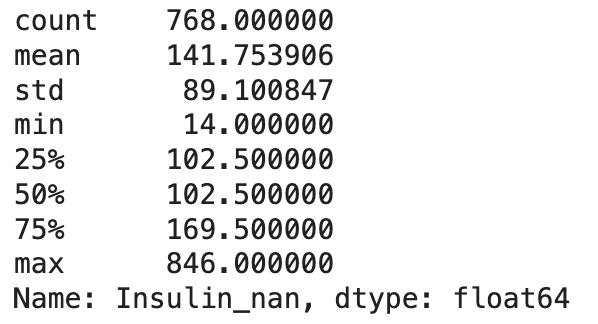
여기서 mean의 값이 중앙값(50%값 = 102.5)에 비해 큰 이유는 최대치가 중앙값에서 매우 멀리 떨어져있어 평균값에 큰 영향을 주었기 때문이다.
그렇기 때문에 이러한 이상치를 없애기 위한 작업을 할 필요가 있다.
IQR3 = df["Insulin_nan"].quantile(0.75)
IQR1 = df["Insulin_nan"].quantile(0.25)
IQR = IQR3 - IQR1
IQROUT = IQR3 + (IQR * 1.5)
OUTOUT 변수는 이 때 270이라는 값을 가지는데 IQR3에 1.5IQR을 뜻한다. 즉 270 이상의 값을 이상치로 판단 후 케이스에서 제거하겠다는 말이다.
df[df["Insulin_nan"] > OUT].shape이때 결과값은 51이 나온다. 제거하려는, 270이상의 값을 가진 케이스가 51개라는 뜻이므로 768개 중 51개를 날리기에는 무리인 것 같아 강의에서는 우선 600이상의 값만 제거하기로 했다.
train = df[df["Insulin_nan"] > 600].shape600 이상의 값을 가지는 케이스는 단 3개로 아주 큰 아웃라이어만 제거한다.
이러한 이상치를 제거한 후 다시 학습을 했을 때 모델의 정확도를 평가했을 때 정확도가 향상되지 않은 것으로 보아 결측치를 제거하는 것이 결과에 항상 좋은 영향을 미치는 것은 아니다 라고 할 수 있겠다. 그래도 유의미한 인사이트를 얻기 위해서는 다양한 방법으로 정확한 데이터를 얻을 수 있도록 하는 것이 좋을 것이다.
3.1.8 피처 스케일링
데이터 스케일링(data scaling)
머신러닝 모델에서 중요한 데이터 전처리 단계 중 하나로, 피처(feature)들마다 데이터값의 범위가 다 제각각이기 때문에 범위 차이가 클 경우 데이터를 갖고 모델을 학습할 때 0으로 수렴하거나 무한으로 발산할 수 있다. 즉, 어떤 피처는 무시되고 어떤 피처는 과도하게 영향을 받을 수 있는 것이다. 따라서 이를 방지하게 위해 스케일링 과정을 통해 모든 피처들의 데이터 분포나 범위를 동일하게 조정할 수 있다.
가장 일반적인 데이터 스케일링 기법에는 다음 4가지가 있다.
- 표준화 (Standardization) : 평균을 빼고 표준편차로 나누어 데이터의 분포를 평균 0, 표준편차 1인 표준 정규 분포로 변환하는 방법이다. 이 방법은 주로 데이터가 정규 분포를 따를 때 사용된다.
- 정규화 (Normalization) : 주어진 범위(예: 0과 1)로 데이터를 조정하는 방법이다. 이 방법은 데이터의 스케일이 제각각일 때 사용됩니다.
- 최소-최대 스케일링 (Min-Max Scaling) : 데이터를 새로운 최소 값과 최대 값 사이의 범위로 조정하는 방법이다. 일반적으로 0과 1사이의 범위로 조정한다.
- 로버스트 스케일링 (Robust Scaling) : 중앙값(median)과 사분위 범위(Interquartile Range, IQR)를 사용하여 이상치에 영향을 덜 받는 방식으로 스케일링하는 방법이다.
우리가 해 볼 스케일링 기법은 1번 표준화 기법이다. 다음 코드를 통해 자세히 알아보자.
# Scikit-learn 라이브러리에서 StandardScaler 클래스를 import
from sklearn.preprocessing import StandardScaler
# StandardScaler 클래스의 인스턴스를 생성하여 scaler 변수에 할당
scaler = StandardScaler()
# fit 메서드를 사용하여 scaler를 주어진 데이터에 fitting
scaler.fit(df[["Glucose", "DiabetesPedigreeFunction"]])하려
# transform 메서드를 사용하여 실제로 데이터를 표준화하여 scale에 저장
scale = scaler.transform(df[["Glucose", "DiabetesPedigreeFunction"]])
scale
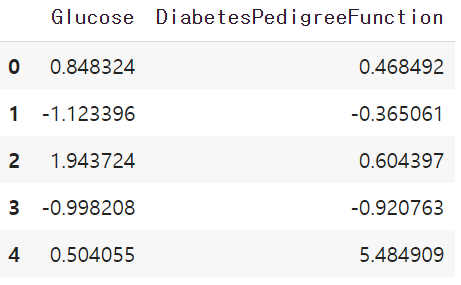
스케일링 한 값을 한것을 "Glucose", "DiabetesPedigreeFunction" 변수에 다시 담아주고 잘 들어갔는지 확인해보자.
df[["Glucose", "DiabetesPedigreeFunction"]] = scale
df[["Glucose", "DiabetesPedigreeFunction"]].head()
히스토그램으로도 한 번 그려보자.
h = df[["Glucose", "DiabetesPedigreeFunction"]].hist(figsize=(15, 3))
* .hist(): 선택된 열에 대해 히스토그램을 그리는 메서드
* figsize=(15, 3): 그림의 크기를 설정
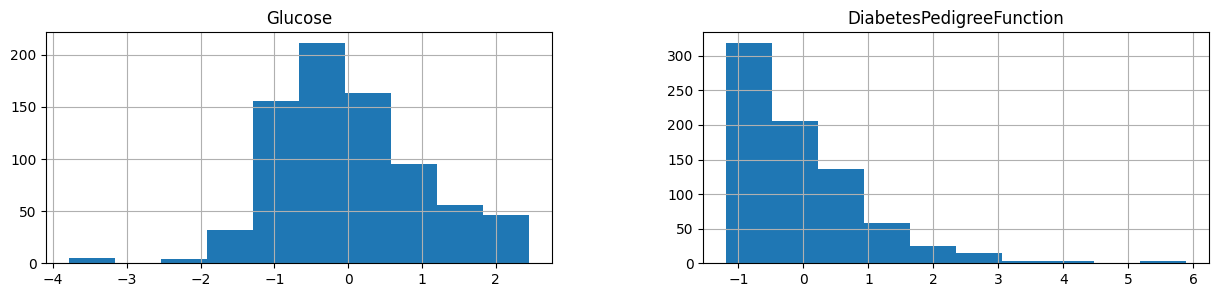
"Glucose"의 범위는 -4부터 2까지 , "DiabetesPedigreeFunction" -1에서 6까지로 범위가 달라짐
이어서 다른 코드들도 계속 실행해주면, 정확도 측정 단계에서 diff_count 값이 15로 나온다.
이 데이터의 경우 수티치 범위의 크기가 크지 않아서 스케일링 작업의 전후의 차이가 크지 않아 보인다. 하지만 수치 범위의 큰 경우 스케일링 기법을 사용하여 예측의 정확도의 차이를 높일 수 있다.
전처리가 예측의 정확도에 큰 영향을 끼칠 수 있고, 모든 전처리 과정이 예측을 정확도를 무조건 높이는 것이 아니기 때문에 EDA를 해보고 feature engineering을 해보고 좋은 성능을 해는지 확인해보는 과정이 필요하다. 좋은 성능을 내지 못할때에는 그 이유가 무엇인지 살펴보는 것도 중요하다.
3.1.9 전처리한 피처를 CSV 파일로 저장하기
스케일링이 별로 성능을 향상시키지 못했기 때문에 주석 처리를 하고 나머지 전처리와 이상치 처리 과정을 다시 실행하낟. 그리고 전처리한 데이터를 CSV 파일에 저장한다.
df.to_csv("data/diabetes_feature.csv")
저장한 데이터를 다시 불러와본다.
pd.read_csv("data/diabetes_feature.csv").head()
불러온 파일을 보면, unnamed: 0이라는 열이 생성된 것을 볼 수 있다. 인덱스로 생성된 열을 함께 저장해서 생긴 것이다. 따라서 위의 csv 파일 저장 코드에 index를 False로 지정하여 index를 없앤 후 다시 CSV 파일에 저장한다.
df.to_csv("data/diabetes_feature.csv", index=False)
그리고 다시 한 번 저장한 데이터를 불러와보면 unnamed: 0 열이 사라진 것을 확인 할 수 있다.
3주차 과제
조원들 모두 정확도를 75% 보다 더 끌어 올리기 위해 많이 노력했지만, 쉽지 않았다.
그래도 모두가 같이 해본 것에 의의를 두기로 했다 : )